Na mídia
Pesquisa Fapesp: Redes brasileiras estão de olho nas variantes do coronavírus
01.07.2022
Frances Jones / Agência Fapesp
Imagem acima: Fotomontagem com imagens do vírus SARS-CoV-2. Crédito: NIAID/RML
Quais são as variantes do vírus causador da Covid-19 em circulação no Brasil no momento e qual é o impacto delas sobre a saúde da população? As novas linhagens do Sars-CoV-2 se propagam com mais facilidade? Causam doença mais ou menos grave? As vacinas e os testes de diagnóstico continuam eficazes? Essas são algumas das perguntas que o trabalho de vigilância genômica pretende responder para dizer quais mutações estão presentes nos vírus em circulação. São os especialistas dessa área que darão o alerta quando surgir uma nova variante de preocupação – aquelas que trazem maior risco potencial à saúde pública –, seja porque é mais transmissível ou letal.
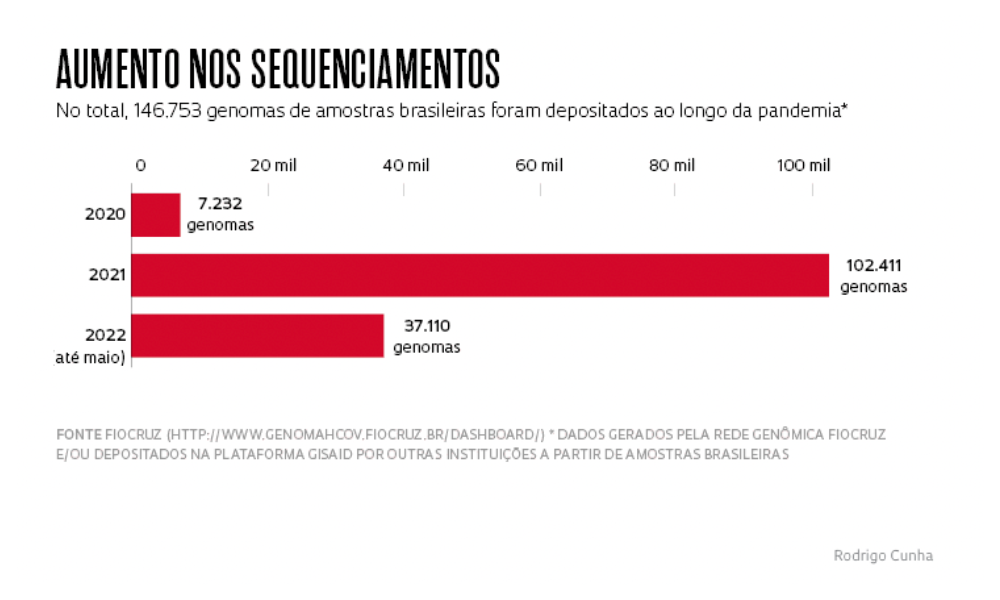
No Brasil, segundo a plataforma internacional Gisaid, apenas 0,47% dos mais de 31 milhões de casos registrados da doença teve amostra sequenciada e, portanto, seu material genético decodificado – bem abaixo dos 5% preconizados por especialistas. Em números absolutos, foram analisados menos de 150 mil genomas. A porcentagem já foi menor, principalmente no primeiro ano da pandemia, por falta de financiamento, equipamentos e insumos. Gisaid é o principal banco internacional de compartilhamento de dados sobre o código genético e as mutações detectadas no novo coronavírus.
Embora os números da vigilância genômica no país tenham melhorado a partir do segundo semestre de 2021 (ver infográfico abaixo), pesquisadores e gestores ainda buscam recursos para garantir a manutenção e a ampliação dos trabalhos. A boa notícia é que as duas grandes redes nacionais de vigilância genômica ganharam recentemente o apoio da Agência de Segurança em Saúde do Reino Unido (UKHSA) para intensificar os trabalhos de sequenciamento genético e identificar possíveis novas variantes em circulação no país.
Pesquisadores da Rede Genômica Fiocruz, apoiada pelo Ministério da Saúde (MS) e coordenada por cientistas da Fundação Oswaldo Cruz (Fiocruz), e da Rede Nacional de Ômicas de Covid-19 (Rede Corona-ômica BR), financiada pelo Ministério da Ciência, Tecnologia e Inovações (MCTI), já participam de reuniões de trabalho com integrantes da plataforma NVAP (New Variant Assessment Platform). O programa foi montado pelo governo britânico no ano passado para reforçar a infraestrutura de outros países e ajudar a encontrar novas variantes. Além do Brasil, Chile, Singapura, Etiópia, Quênia, Nigéria, Paquistão e Trinidad e Tobago fazem parte da iniciativa.
"O suporte da agência do governo britânico não é o único, mas é um apoio importante para continuarmos com nossas análises e podermos gerar informação genômica", destaca a virologista Marilda Siqueira, chefe do Laboratório de Vírus Respiratórios e do Sarampo do Instituto Oswaldo Cruz (IOC/Fiocruz). A unidade, um centro de pesquisa, desenvolvimento e formação na área de vírus que afetam o sistema respiratório, serve de referência em Covid-19 para o MS, no Brasil, e para a Organização Mundial da Saúde (OMS), nas Américas.
À frente da Rede Genômica Fiocruz, Siqueira conta que o suporte britânico poderá fazer com que mais unidades da Fiocruz passem a realizar análises genômicas do Sars-CoV-2. Hoje, dos 11 centros da instituição, apenas oito fazem esse tipo de trabalho nos estados do Amazonas, Ceará, Pernambuco, Piauí, Bahia, Rio de Janeiro, Minas Gerais e Paraná.
"Realizar análise genômica é uma atividade cara e complexa. Exige não apenas pessoal qualificado, mas equipamentos e insumos dispendiosos", explica a pesquisadora. De acordo com ela, o apoio britânico, estimado para durar um ano, prevê, no caso da rede Fiocruz, o fornecimento de insumos e de treinamento de pessoal, nas áreas de bioinformática, análise de dados e vigilância epidemiológica. "Começamos a receber os insumos em abril e já realizamos alguns treinamentos."
Na outra frente de apoio ao Brasil, coordenada pela Rede Corona-ômica BR, o acordo com os britânicos foi selado um pouco mais tarde e, até o final de junho, os insumos ainda não haviam chegado, embora os especialistas do Reino Unido já estivessem realizando workshops com a equipe. Formada em 2020, a rede agrupa 19 núcleos de pesquisa, principalmente de universidades. "A ideia dos britânicos é apoiar redes já estabelecidas e ceder insumos para que possamos aprofundar os esforços de sequenciamento genômico e de vigilância", diz o virologista Fernando Spilki, coordenador da rede e professor da Universidade Feevale, em Nova Hamburgo (RS).
"Para nós, o mais relevante em relação a esse projeto é a formação de pessoal. O Reino Unido tem uma grande expertise no cruzamento dos dados de vigilância genômica com informações epidemiológicas", afirma o pesquisador. "Pode parecer muito fácil correlacionar uma determinada variante ou sublinhagem a um surto ou elevação de casos, mas muitos aspectos precisam ser considerados nessa análise, como questões sociais, geográficas, econômicas e temporais."
Um dos projetos no âmbito da Rede Corona-ômica apoiados pela plataforma NVAP é liderado pela médica Ester Sabino. Professora do Instituto de Medicina Tropical (IMT) da Faculdade de Medicina da Universidade de São Paulo (FM-USP), ela é a coordenadora do Centro Conjunto Brasil-Reino Unido para Descoberta, Diagnóstico, Genômica e Epidemiologia de Arbovírus (Cadde), financiado pela FAPESP e pela agência britânica de fomento à pesquisa em saúde Medical Research Council (ver Pesquisa FAPESP no 304).
Em 2021, Sabino deu início a um projeto de vigilância genômica com o apoio da empresa de varejo Magalu, no âmbito de uma iniciativa do Grupo Mulheres do Brasil. A proposta inicial era montar em cada capital brasileira um grupo que trabalhasse em rede e sequenciasse mensalmente 100 amostras do vírus da Covid-19.
Os recursos chegaram em novembro, contratamos as coordenadoras, recebemos os primeiros reagentes e iniciamos o treinamento do pessoal", conta Sabino. A iniciativa chegou a 19 capitais, inclusive de estados distantes do Centro-sul do país, como Amapá, Acre e Rondônia, que raramente são contemplados em projetos de grande porte porque carecem de infraestrutura, informa a pesquisadora. Alguns laboratórios já estão fazendo as análises genômicas. O acerto inicial com o grupo empresarial prevê recursos para seis meses de trabalho.
"Essa iniciativa do Mulheres do Brasil é importante porque está capacitando grupos que nunca tiveram experiência com sequenciamento", comenta o virologista Renato Santana, do Departamento de Genética, Ecologia e Evolução do Instituto de Ciências Biológicas da Universidade Federal de Minas Gerais (ICB-UFMG), cujo laboratório integra o guarda-chuva da Rede Corona-ômica. "Eles estão recebendo equipamentos e insumos para fazer o sequenciamento e o monitoramento do Sars-CoV-2 em seus próprios locais."
O laboratório chefiado por Santana também receberá reagentes dentro da plataforma NVAP, assim como os laboratórios contemplados pelo projeto do Mulheres do Brasil e outros da Rede Corona-ômica. "A ideia é sequenciar pelo menos 5 mil amostras por mês nas cinco regiões do Brasil", afirma o pesquisador.
Um parceiro importante para a execução do projeto, conta Santana, é o grupo Hermes Pardini, com sede em Belo Horizonte, cuja rede tem mais de 6 mil laboratórios de análises clínicas parceiros no país. "Todas as 16 mil amostras positivas de Covid diagnosticadas pelo grupo a partir de janeiro deste ano já estão no meu laboratório. Só estamos esperando os reagentes chegarem para redirecionar para os laboratórios que vão fazer o sequenciamento", afirma.
Vigilância no Brics
A falta de amostras no país para a realização de sequenciamentos é um desafio para quem trabalha com vigilância genômica, conforme explica a bioinformata Ana Tereza Ribeiro de Vasconcelos, coordenadora do Laboratório de Bioinformática (Labinfo) do Laboratório Nacional de Computação Científica (LNCC), unidade de pesquisa do MCTI. "Tem sido difícil obter amostras. Há pouca testagem atualmente, pois as pessoas pararam de realizar ensaios moleculares pela técnica RT-PCR, que geram amostras do vírus, e passaram a fazer testes de antígenos nas farmácias ou nos laboratórios, que não produzem amostras", diz a pesquisadora.
Vasconcelos está à frente de outra rede de vigilância genômica que integra uma cooperação internacional com os demais países do Brics (além do Brasil, também a Rússia, Índia, China e África do Sul). A proposta inicial da cooperação, que começou em 2021 e está prevista para se encerrar ao final deste ano, prevê o sequenciamento de 3 mil amostras em cada uma das cinco nações, com trocas de informações e análises conjuntas. Outra iniciativa no país é coordenada pelo Instituto Todos pela Saúde, apoiado pela Fundação Itaú Unibanco, que dispunha em 2021 de R$ 200 milhões para investir em vigilância genômica.
"Estou sequenciando agora amostras de janeiro a março que recebi de laboratórios parceiros", relata a bioinformata. Os genomas produzidos no Brasil dentro desse programa são depositados no Gisaid e no banco de dados da China, este último um dos três mais importantes do mundo. O coordenador geral do projeto é o bioinformata brasileiro Tulio de Oliveira, que está à frente dos laboratórios do Centro para Resposta Epidêmica e Inovação (Ceri), da Universidade de Stellenbosch, e do KwaZulu-Natal Research and Innovation Sequencing Platform (Krisp), ambos na África do Sul.
"Esse vírus sofre mutações muito rapidamente e é de alta transmissibilidade", diz Vasconcelos. "É difícil sincronizar as ondas de infecção que acontecem nos diferentes lugares do mundo. Mas estamos acompanhando o que ocorre em cada país, e nas reuniões mensais com pesquisadores dos outros países já sabemos de antemão quais as variantes estão em circulação em um lugar e outro. Esse trabalho é importante e pode nos trazer alguma vantagem no combate ao vírus."
As filhas da ômicron
Diferentes sublinhagens da variante tornam-se dominantes
Desde o fim de 2021, quando foi detectada pela primeira vez na África do Sul, a variante ômicron foi ganhando espaço entre as demais linhagens do Sars-CoV-2 e tornou-se a dominante no mundo (ver Pesquisa FAPESP no 311). Em junho, era a única a receber a denominação de variante de preocupação (VOC) pela Organização Mundial da Saúde (OMS). As cepas mais antigas (delta, alfa, beta e gama) entraram em outra categoria, a das "VOC anteriormente em circulação".
Em junho, pelo menos cinco subvariantes, descendentes das primeiras cepas da ômicron, chamavam a atenção de pesquisadores. A Rede Genômica Fiocruz indicava em seu site que 48% dos genomas de Sars-CoV-2 produzidos em maio no Brasil eram da subvariante BA.2, um pouco mais transmissível do que a ômicron original; 16% eram da BA.1; e outros 15% da BA.1.1.
No mundo, as linhagens de BA.2 ainda eram as dominantes, mas já apresentavam queda. Subindo em prevalência estavam as variantes BA.2.12.1 (responsável pela maioria dos casos nos Estados Unidos), BA.5 (dominante em Portugal) e BA.4 (detectada na África do Sul e em outros 41 países). As três carregam em seu material genético mutações que parecem conferir maior transmissibilidade e, ao mesmo tempo, torná-las imunes aos anticorpos gerados pela vacinação ou infecções anteriores.
"Está acontecendo agora no Brasil algo similar ao que ocorreu em 2021 com a delta. Enquanto ela perdia força no resto do mundo, estava chegando aqui", diz o virologista Fernando Spilki, coordenador da Rede Corona-ômica BR."Em muitos estados já estamos vendo há um bom tempo o predomínio da BA.2; em outros estamos começando a ver o crescimento da BA.4 e da BA.5. A nossa epidemia continua sendo um tanto diversificada quanto à circulação de diferentes linhagens, comparada com a dinâmica vista em outros países."
Projeto
Centro Conjunto Brasil-Reino Unido para Descoberta, Diagnóstico, Genômica e Epidemiologia de Arbovírus (Cadde) (nº 18/14389-0 ); Modalidade: Projeto Temático; Acordo: MRC, UKRI; Pesquisadora responsável: Ester Cerdeira Sabino (USP); Investimento: R$ 8.640.246,96.
Artigo científico
BRITO, A. F. et al. Global disparities in Sars-CoV-2 genomic surveillance. Medrxiv. 9 dez. 2021.
Este texto foi originalmente publicado por Pesquisa FAPESP de acordo com a licença Creative Commons CC-BY-NC-ND.
Conteúdo reproduzido na íntegra da Agência Fapesp (clique aqui)







