Monitoramento das variantes do SARS-CoV-2 - relatório 6
DATA DE PUBLICAÇÃO: 13.12.2021
Semanas 43 a 48
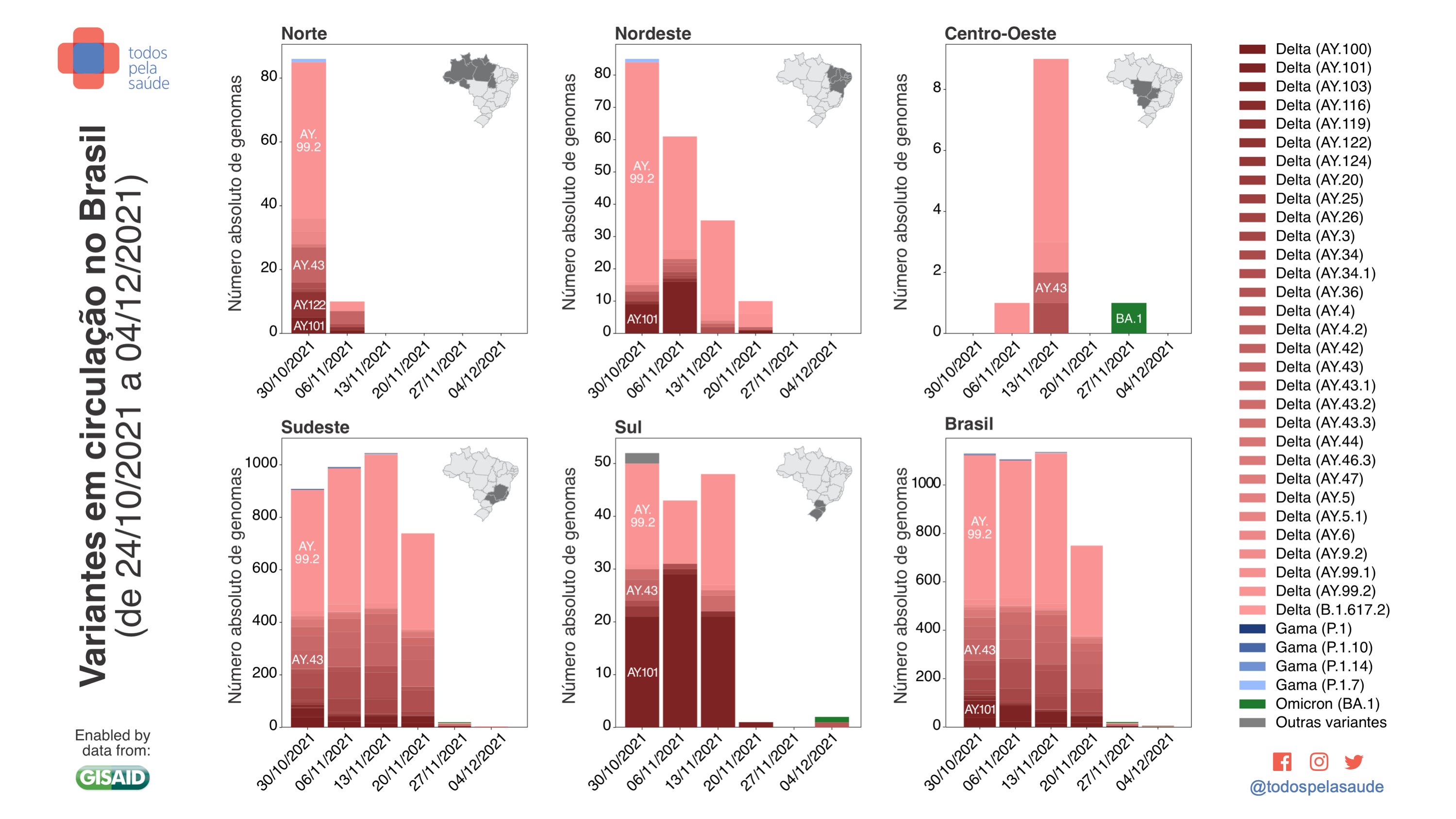
Variantes detectadas nas cinco regiões brasileiras, de 24/10 a 04/12/21, com 4.152 genomas sequenciados por laboratórios independentes:
- Delta = 4.130 (99,47%)
- Gama = 11 (0,26%)
- Ômicron = 5 (0,12%)
- Outras = 6 (0,14%)
As análises são do pesquisador científico Anderson Brito, do Instituto Todos pela Saúde (ITpS).
Até 11/12, cinco casos da variante Ômicron foram detectados no Brasil e confirmados por sequenciamento. Até então, todos são casos importados:
- São Paulo - 3 casos
- Distrito Federal - 1 caso
- Rio Grande do Sul - 1 caso
Para outras informações, clique aqui.
 Variantes Ômicron BA.1 tem uma deleção no gene da proteína spike. Com testes RT-PCR especiais é possível identificar amostras com essa deleção e rapidamente confirmar casos da Ômicron por sequenciamento. Isso é positivo por um lado, mas por outro leva a vieses de amostragem.
Variantes Ômicron BA.1 tem uma deleção no gene da proteína spike. Com testes RT-PCR especiais é possível identificar amostras com essa deleção e rapidamente confirmar casos da Ômicron por sequenciamento. Isso é positivo por um lado, mas por outro leva a vieses de amostragem.
Agora que amostras da Ômicron serão preferencialmente sequenciadas, a amostragem em todo o mundo será enviesada, e nossa percepção da real prevalência da variante será prejudicada. Com essa ressalva, neste relatório apresentamos as proporções de variantes detectadas no Brasil.
Principais variantes detectadas no Brasil em cada semana (datas = último dia das semanas). No mapa, o tamanho dos círculos (pizzas) denotam a quantidade de genomas amostrados em cada estado e as cores indicam as variantes (para Ômicron, leia as ressalvas acima).
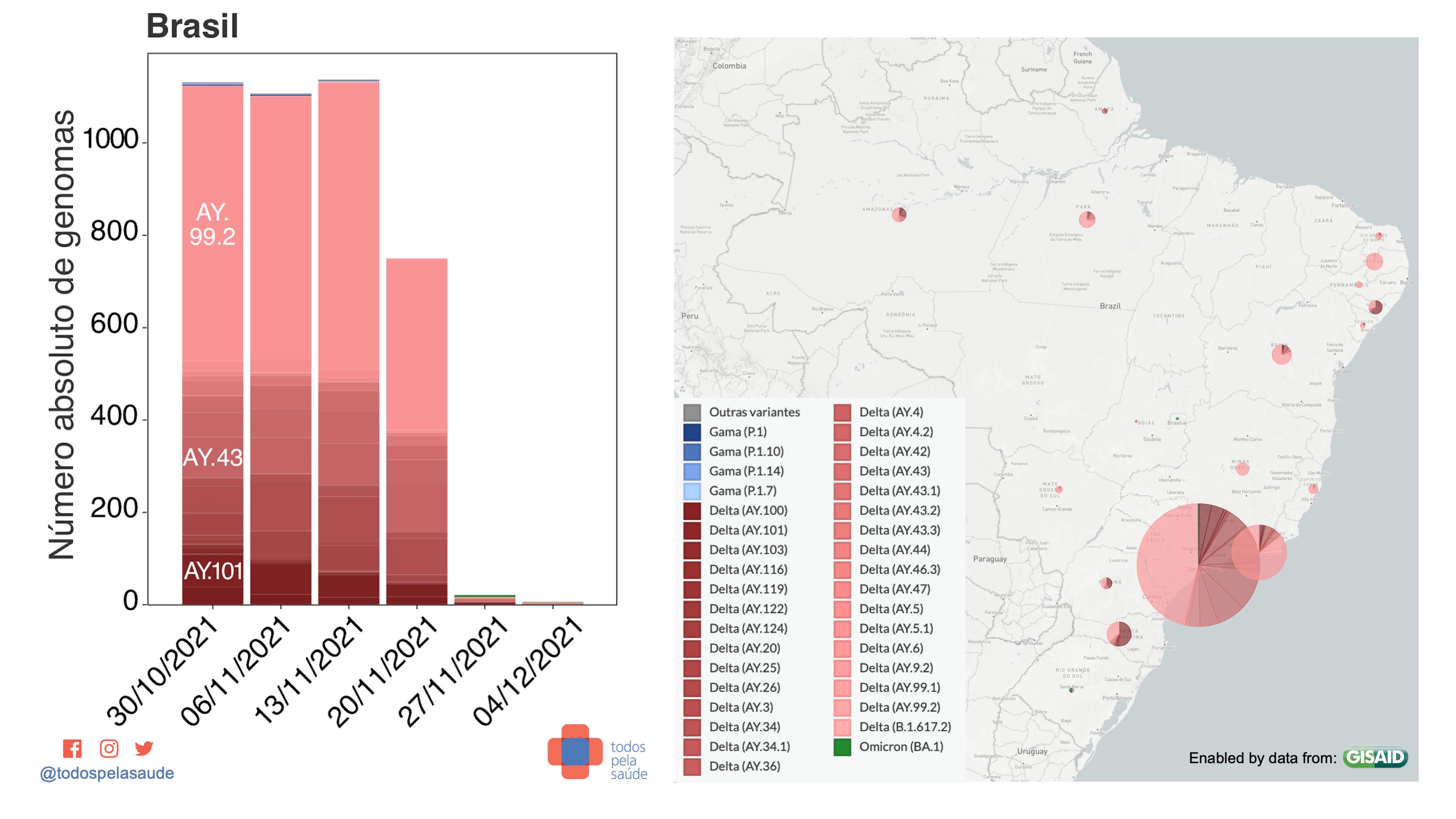
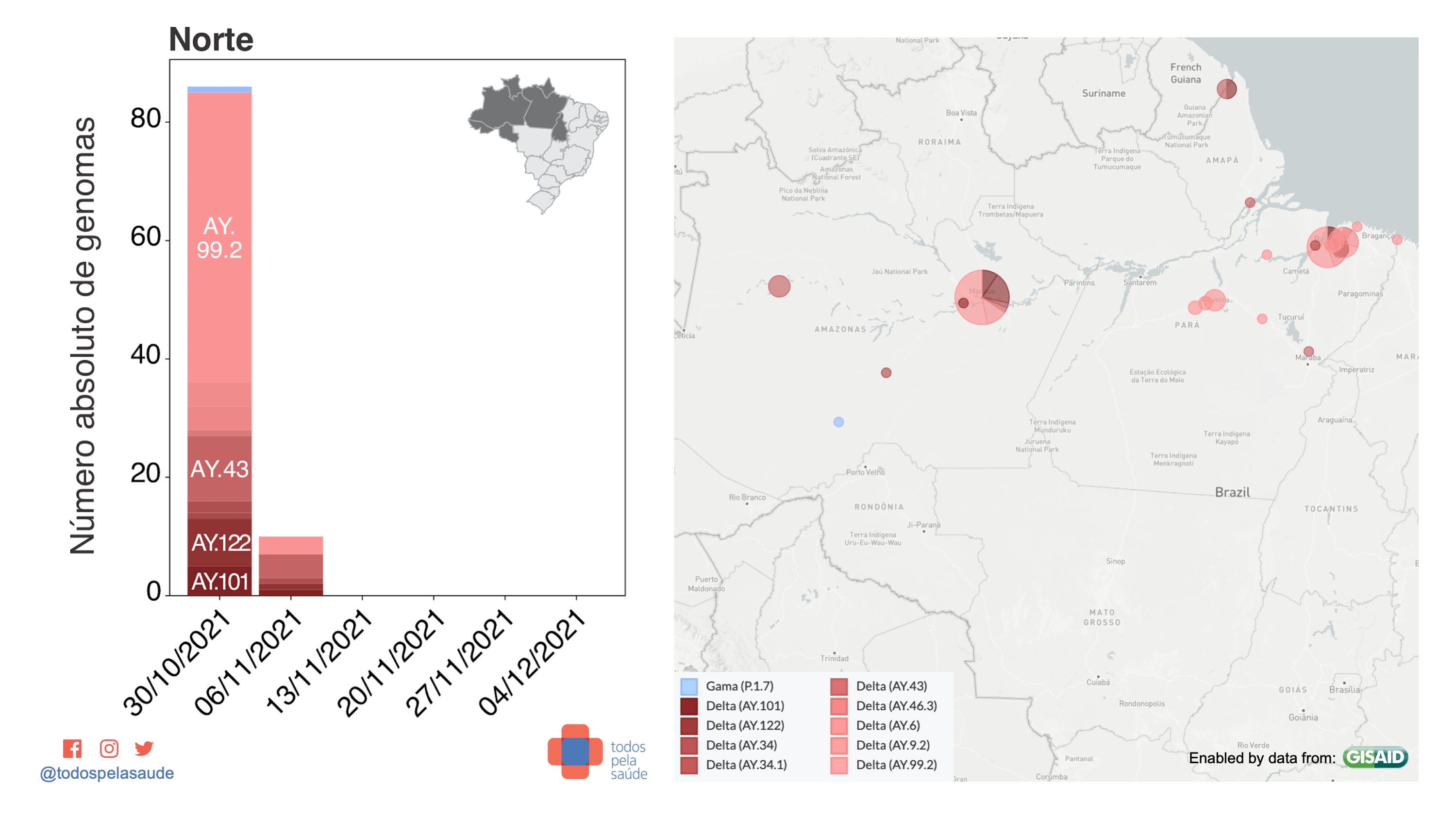
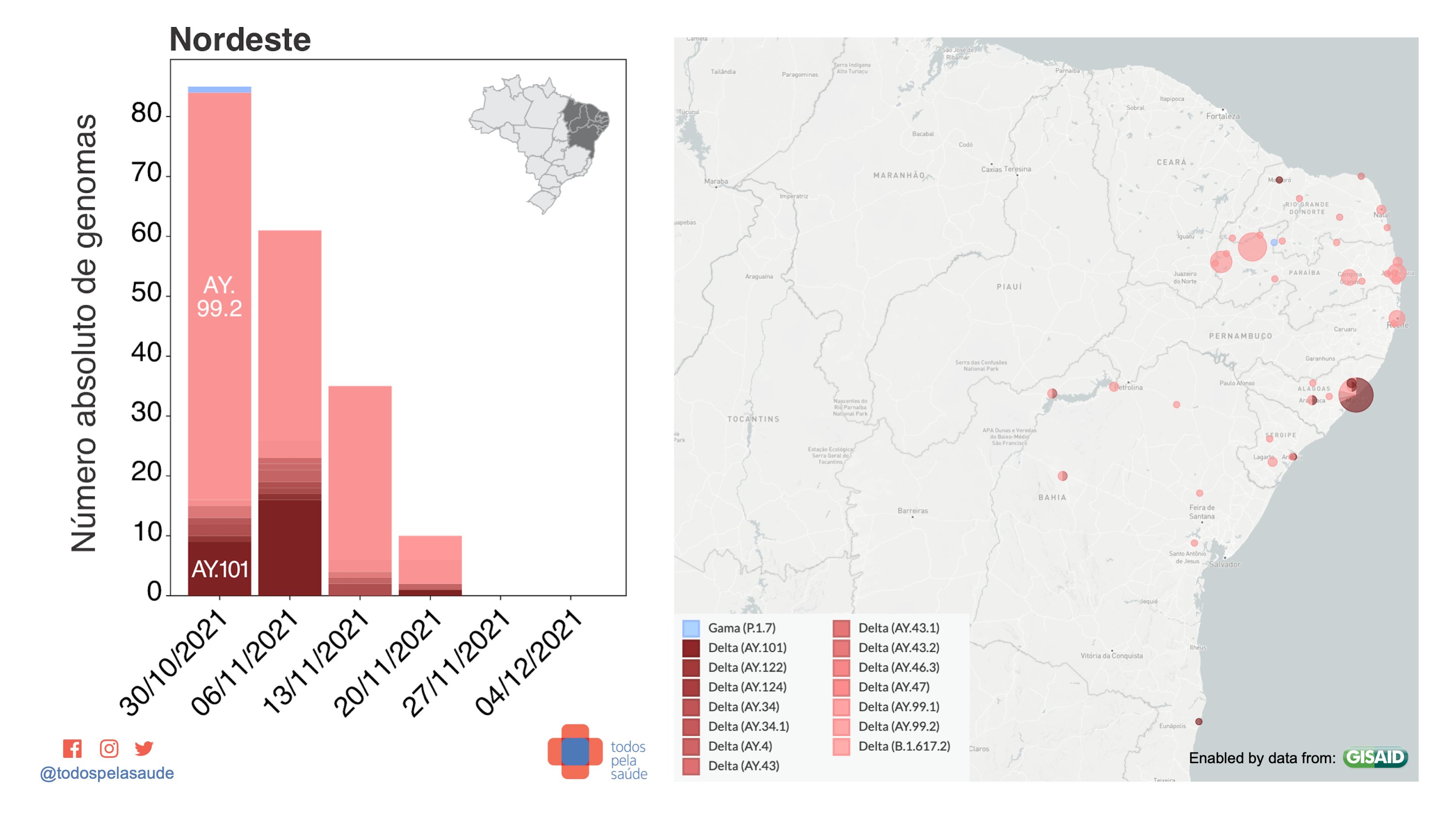
Nos mapas abaixo, vemos os municípios de origem de genomas de variantes sequenciadas nas últimas semanas. Apenas genomas submetidos com o nome da cidade de origem são mostrados no mapa. Para saber sobre a cobertura estadual, veja o mapa acima.
Dados de vigilância genômica da região Norte. 96 genomas sequenciados por dois laboratórios:
- Delta = 95 (98,96%)
- Gama = 1 (1,04%)
- Ômicron = 0
- Outras = 0
Com dados de: Fiocruz (RJ/AM)
 Dados de vigilância genômica da região Nordeste. 191 genomas sequenciados por quatro laboratórios:
Dados de vigilância genômica da região Nordeste. 191 genomas sequenciados por quatro laboratórios:
- Delta = 190 (99,48%)
- Gama = 1 (0,52%)
- Ômicron = 0
- Outras = 0
Com dados de: Dasa, Fiocruz (RJ/PE) e LACEN/BA
Dados de vigilância genômica da região Centro-Oeste. 11 genomas sequenciados por dois laboratórios:
- Delta = 10 (90,91%)
- Gama = 0
- Ômicron = 1 (9,09%)
- Outras = 0
Com dados de: Dasa e Adolfo Lutz
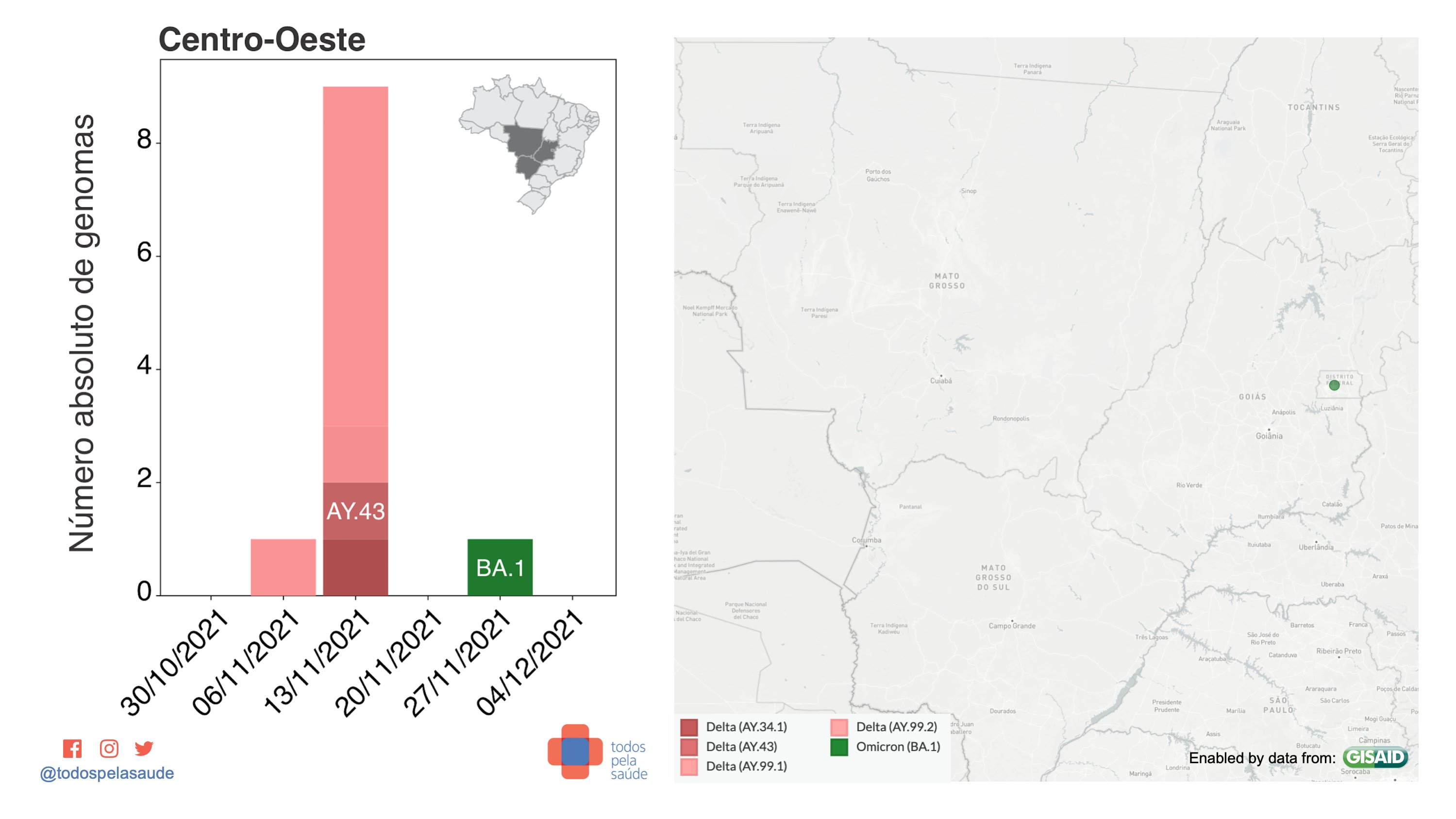
Dados de vigilância genômica da região Sudeste. 3.708 genomas sequenciados por oito laboratórios:
- Delta = 3.692 (99,57%)
- Gama = 9 (0,24%)
- Ômicron = 3 (0,08%)
- Outras = 4 (0,11%)
Com dados de: Butantan, LNCC, Dasa, Fiocruz (RJ), UNESP, Hospital Israelita Albert Einstein e Adolfo Lutz
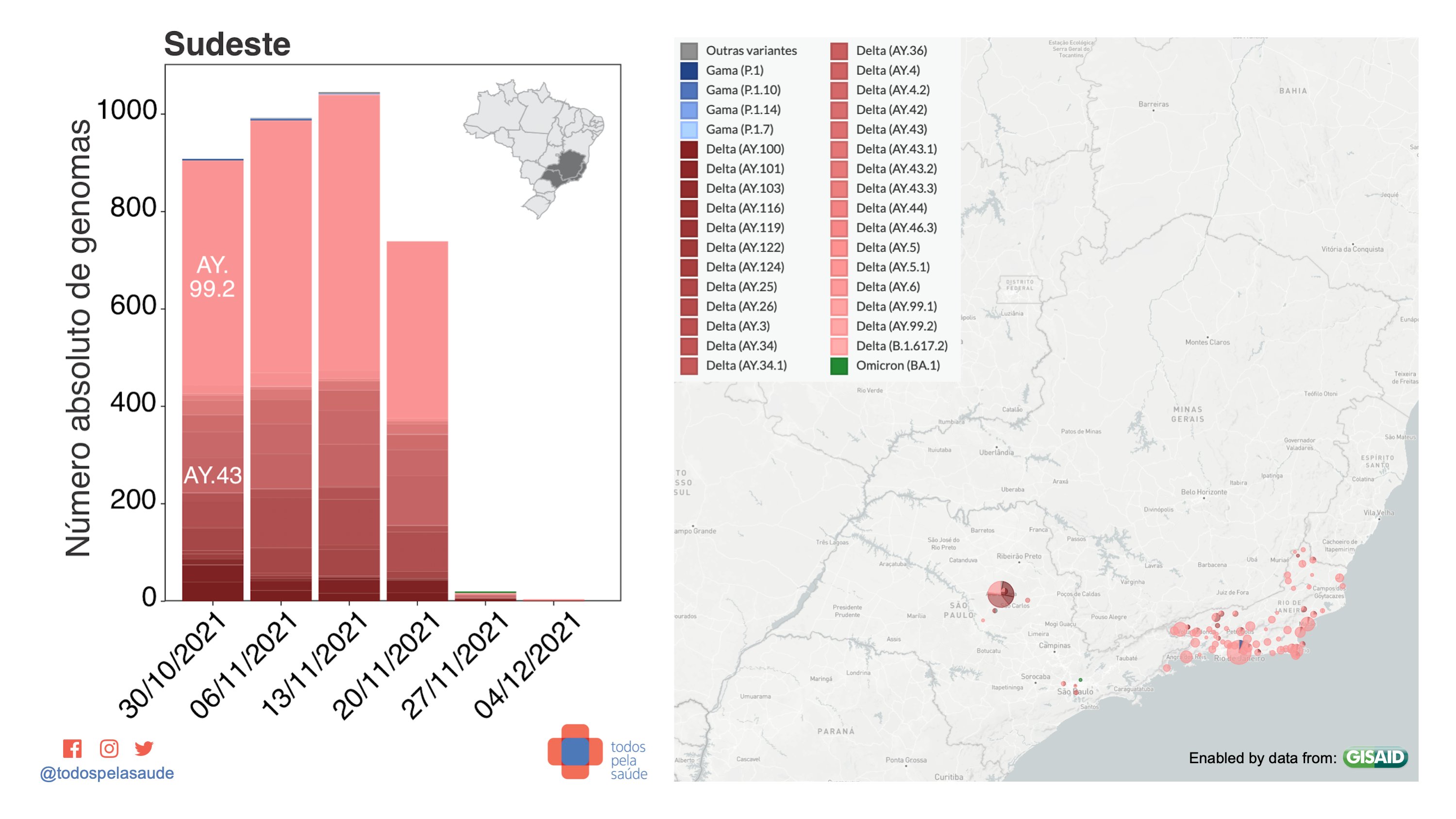
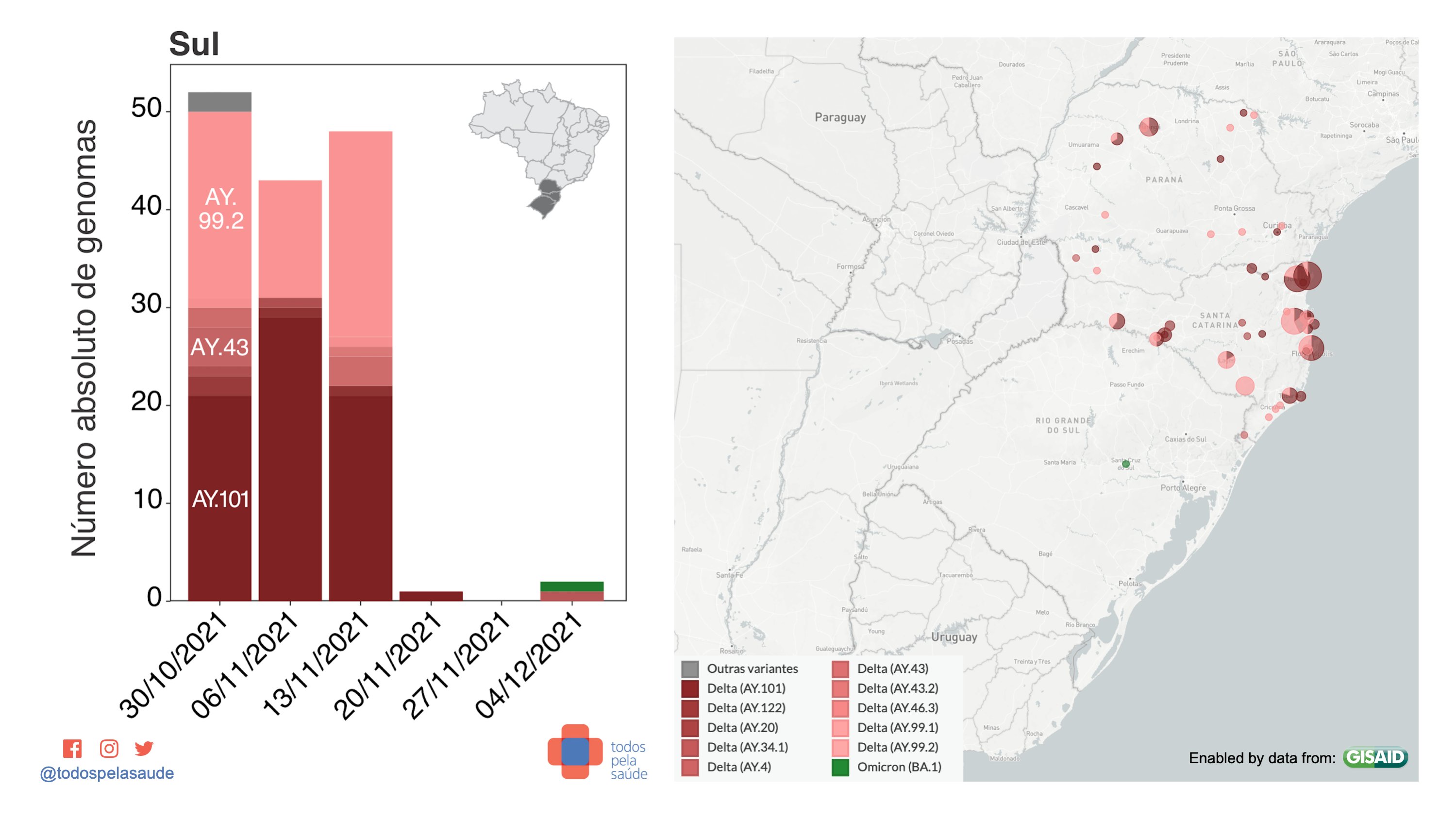
Dados de vigilância genômica da região Sul. 146 genomas sequenciados por quatro laboratórios:
- Delta = 143 (97,95%)
- Gama = 0
- Ômicron = 1 (0,68%)
- Outras = 2 (1,37%)
Com dados de: Fiocruz (RJ/AM), Feevale e CDCT
Para navegar, filtrar e interagir com algumas das informações apresentadas acima, clique aqui. O ITpS agradece aos pesquisadores e laboratórios que coletaram, geraram e submeteram os dados e metadados, aqui apresentados, para o banco de dados Gisaid.
We gratefully acknowledge all data contributors, i.e. the Authors and Originating labs, responsible for obtaining the specimens, and their Submitting labs for generating the genetic sequence and metadata and sharing via the GISAID Initiative (*), on which this research is based.
* Elbe, S., and Buckland -Merrett, G. (2017) Data, disease and diplomacy: GISAID's innovative contribution to global health. Global Challenges, 1:33-46. DOI: 10.1002/gch2.1018https://onlinelibrary.wiley.com/doi/10.1002/gch2.1018
VOLTAR

 Variantes Ômicron BA.1 tem uma deleção no gene da proteína spike. Com testes RT-PCR especiais é possível identificar amostras com essa deleção e rapidamente confirmar casos da Ômicron por sequenciamento. Isso é positivo por um lado, mas por outro leva a vieses de amostragem.
Variantes Ômicron BA.1 tem uma deleção no gene da proteína spike. Com testes RT-PCR especiais é possível identificar amostras com essa deleção e rapidamente confirmar casos da Ômicron por sequenciamento. Isso é positivo por um lado, mas por outro leva a vieses de amostragem. Dados de vigilância genômica da região Nordeste. 191 genomas sequenciados por quatro laboratórios:
Dados de vigilância genômica da região Nordeste. 191 genomas sequenciados por quatro laboratórios:


