Monitoramento das variantes do SARS-CoV-2 - relatório 7
DATA DE PUBLICAÇÃO: 25.01.2022
Semanas 49 (2021) a 2 (2022)
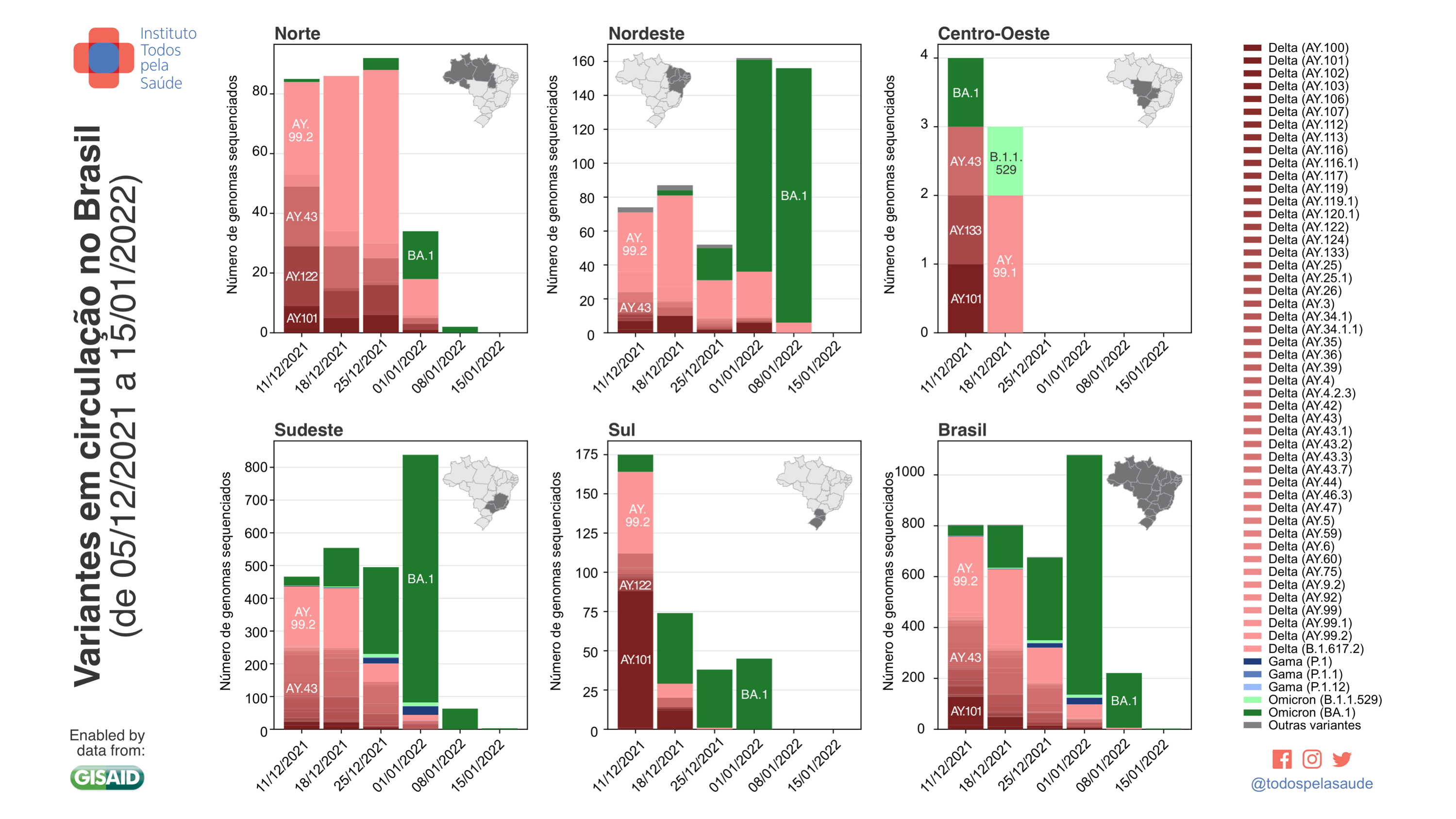
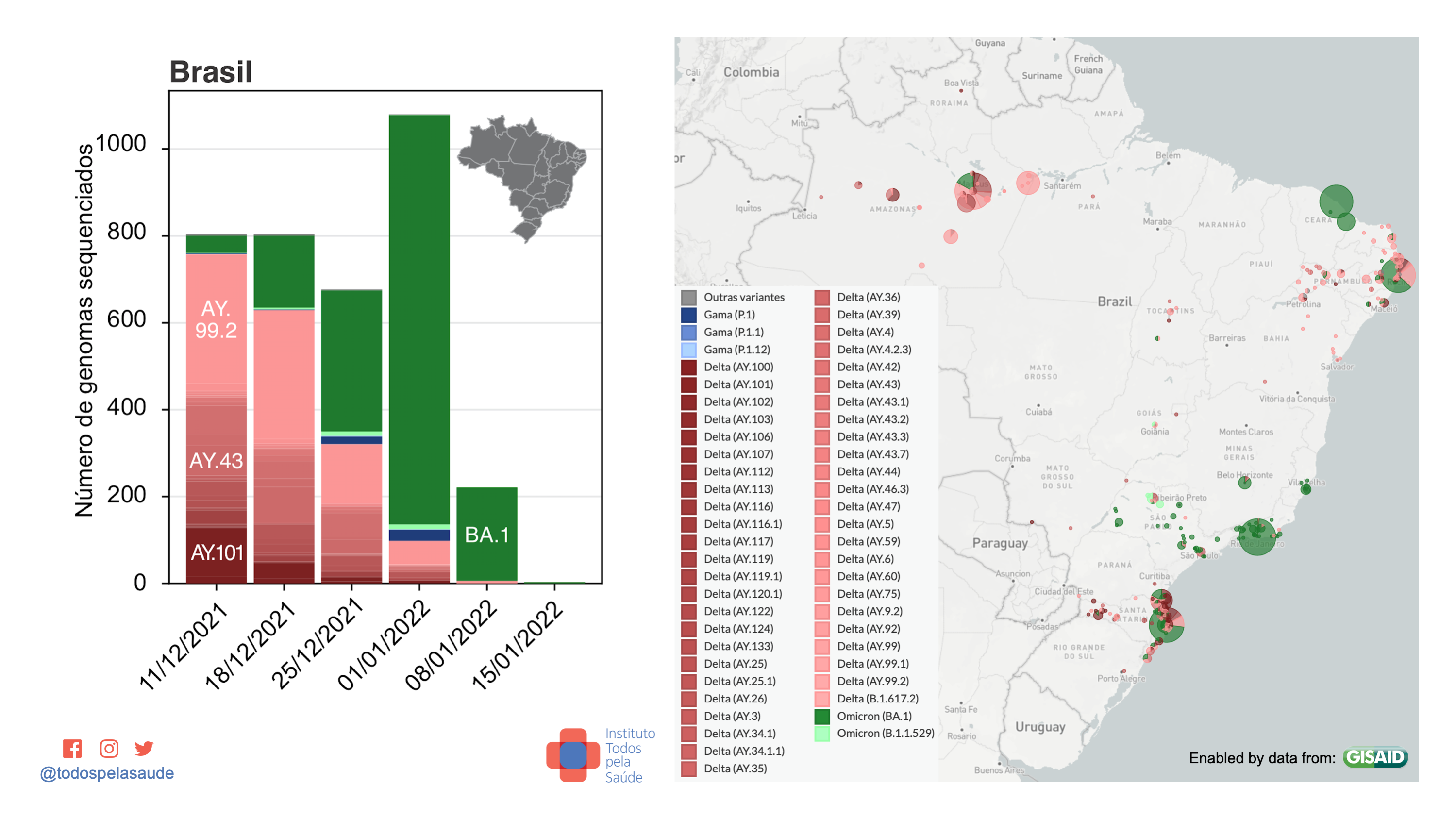
Variantes detectadas no Brasil, de 5/12/21 a 15/1/2022, com 3.589 genomas sequenciados por laboratórios independentes:
- Ômicron = 1.718 (47,87%)
- Delta = 1.812 (50,49%)
- Gama = 50 (1,39%)
- Outras = 9 (0,25%)
As análises são do pesquisador científico Anderson Brito, do Instituto Todos pela Saúde (ITpS).
Na Europa e Ásia, a sublinhagem BA.2 da variante Ômicron vem substituindo a principal sub-linhagem em circulação, a BA.1. Pesquisadores ainda buscam entender se a BA.2 é mais transmissível que a BA.1. Até hoje ainda não há registros da BA.2 no Brasil.
Nos painéis abaixo, vemos as proporções de cada sublinhagem das principais variantes em circulação no Brasil. Tais proporções devem ser avaliadas com cautela, pois não sabemos ao certo quais estratégias de amostragem foram usadas. Logo, existem vieses.
Número de genomas sequenciados por semana (datas = último dia das semanas). No mapa, o tamanho dos círculos (pizzas) denotam a quantidade de genomas amostrados nos estados e as cores indicam as variantes.
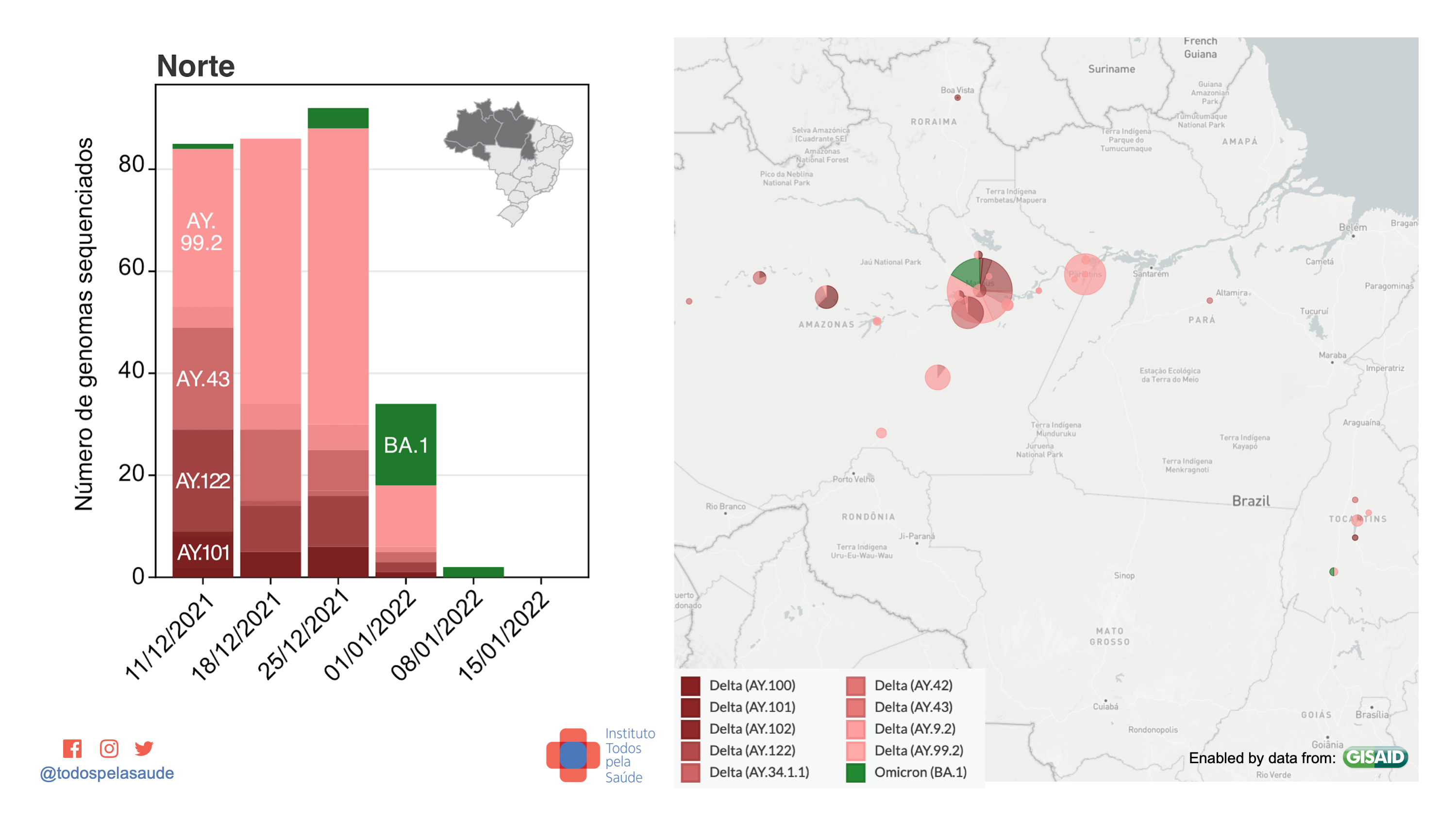
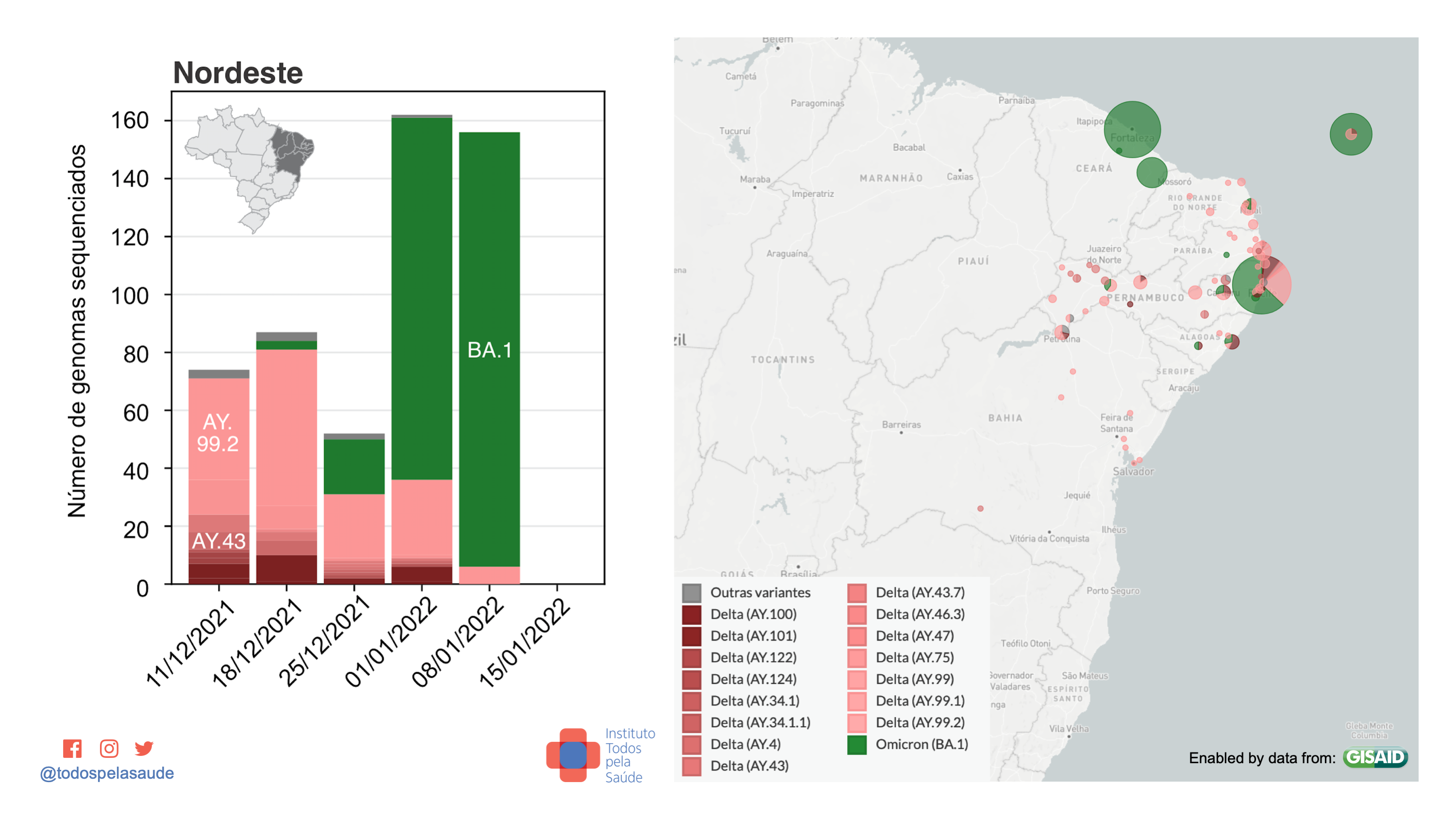
Nos mapas abaixo, vemos os municípios de origem de genomas de variantes sequenciadas nas últimas semanas. Apenas genomas submetidos com o nome da cidade de origem são mostrados no mapa. Para saber sobre a cobertura estadual, veja o primeiro mapa (acima).
Dados de vigilância genômica da região Norte. 299 genomas sequenciados por três laboratórios:
- Ômicron = 23 (7,69%)
- Delta = 276 (92,31%)
- Gama = 0
- Outras = 0
Com dados de: Fiocruz/RJ, Fiocruz/AM e UFT
Dados de vigilância genômica da região Nordeste. 531 genomas sequenciados por quatro laboratórios:
- Ômicron = 297 (55,93%)
- Delta = 225 (42,37%)
- Gama = 0
- Outras = 9 (1,69%)
Com dados de: Fiocruz/RJ, Fiocruz/PE, Fiocruz/CE e Lacen/BA
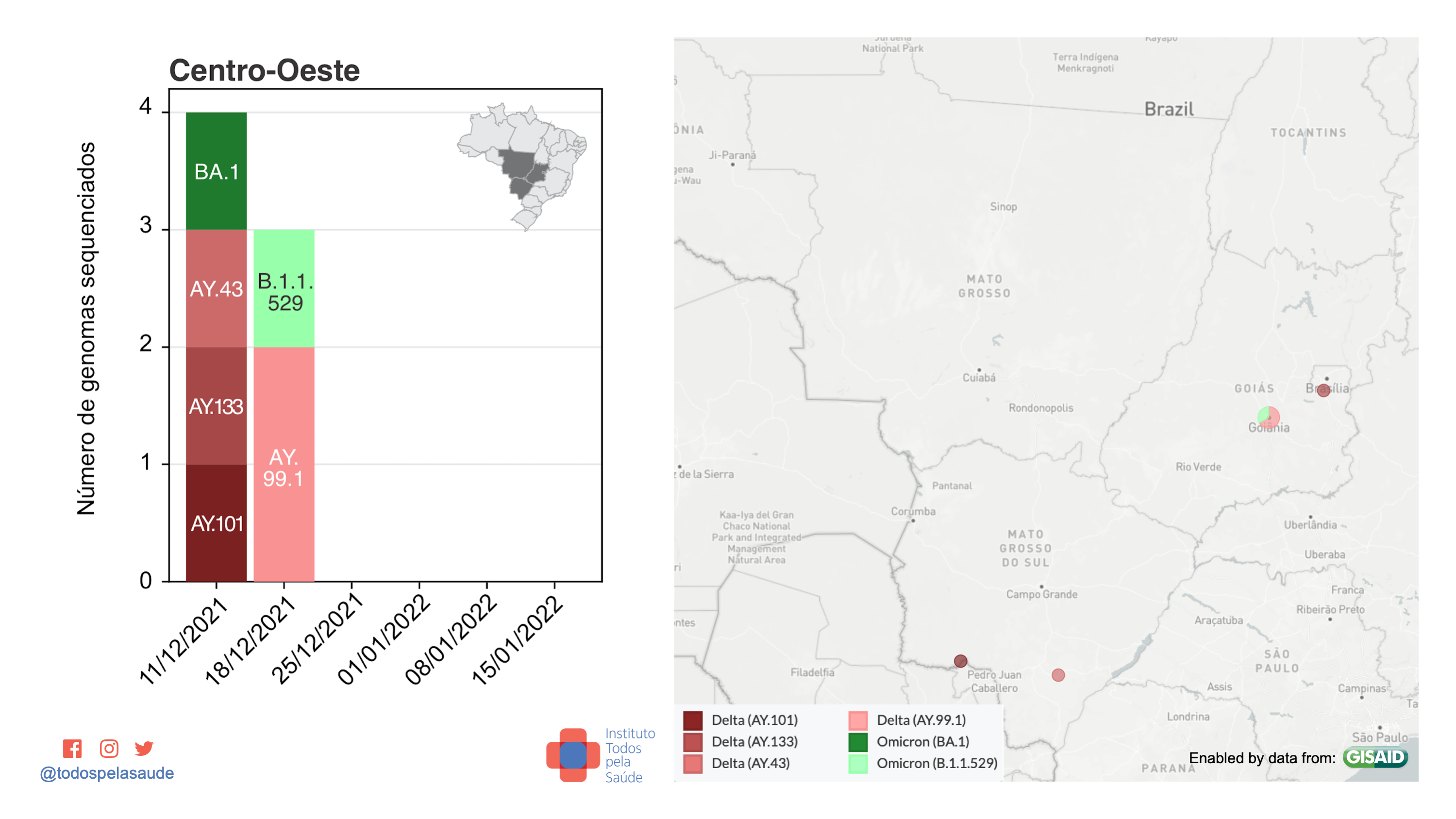
Dados de vigilância genômica da região Centro-Oeste. 7 genomas sequenciados por quatro laboratórios:
- Ômicron = 2 (28,57%)
- Delta = 5 (71,43%)
- Gama = 0
- Outras = 0
Com dados de: Fiocruz/RJ, Adolfo Lutz, Dasa e UFG
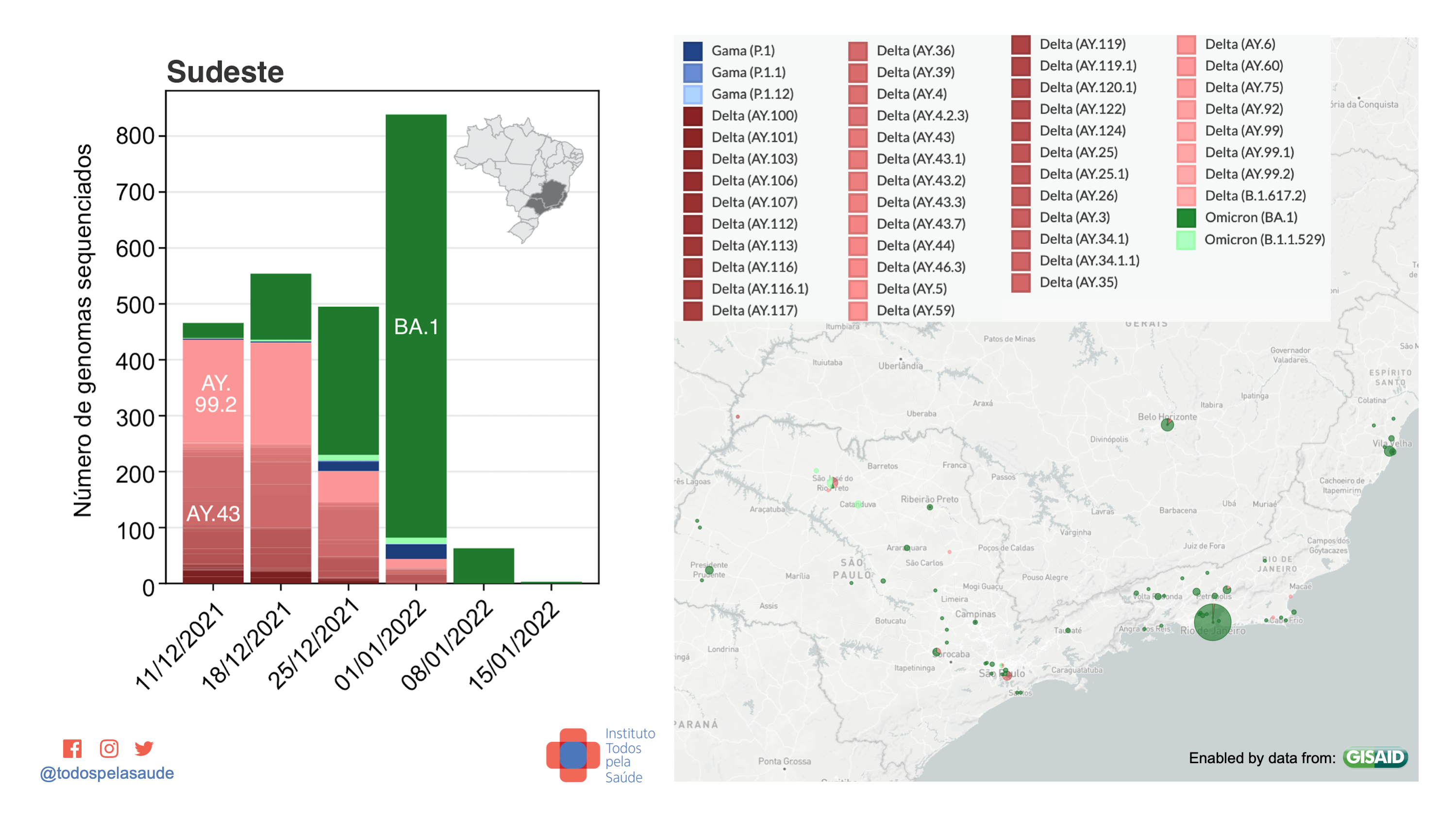
Dados de vigilância genômica da região Sudeste. 2.420 genomas sequenciados por dez laboratórios:
- Ômicron = 1.258 (51,98%)
- Delta = 1.112 (45,95%)
- Gama = 50 (2,07%)
- Outras = 0
Dados: Butantan, Fiocruz/RJ, Adolfo Lutz, Albert Einstein, Famerp, Funed, FMABC, Dasa, Unesp e IMT
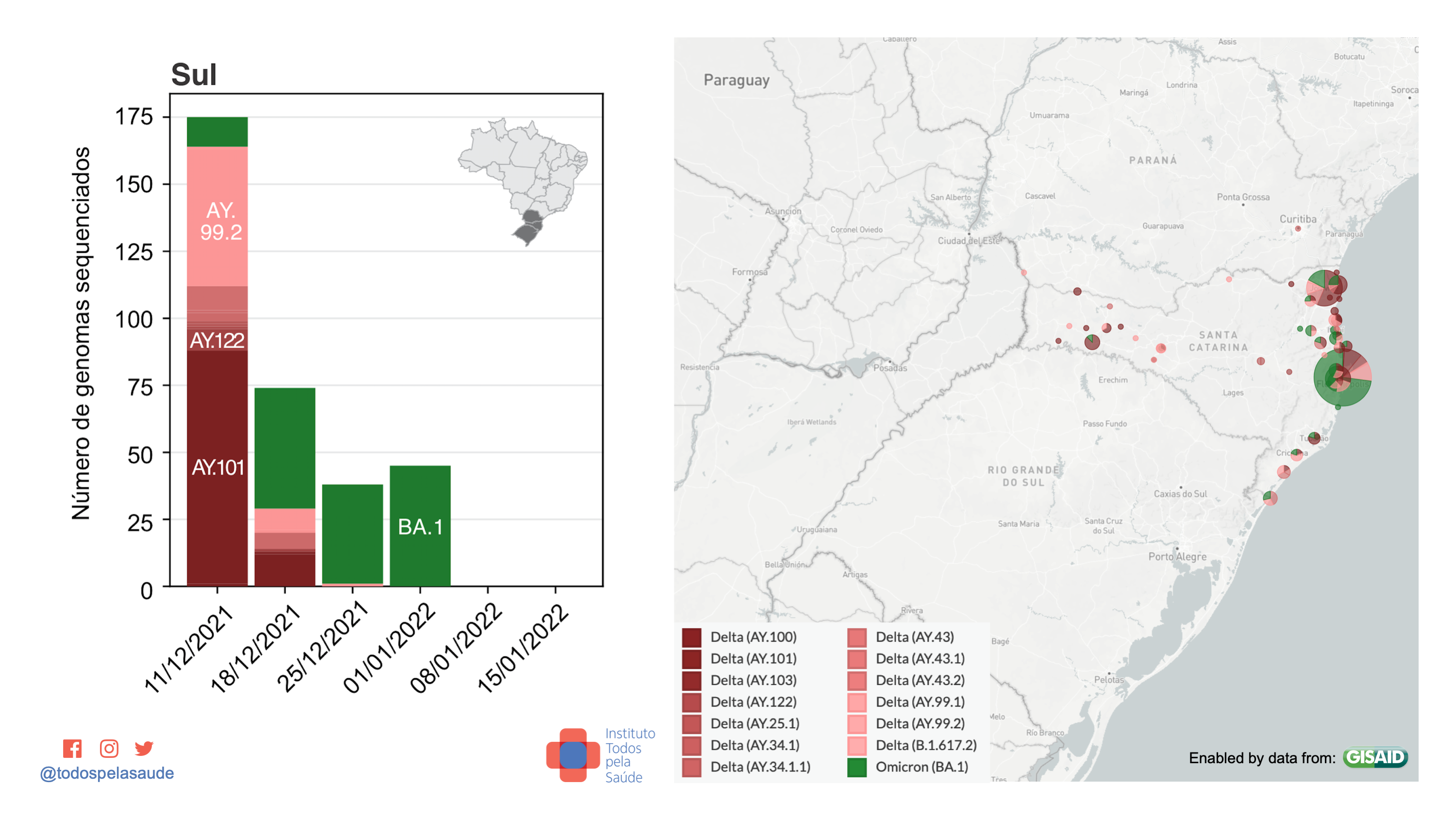
Dados de vigilância genômica da região Sul. 332 genomas sequenciados por quatro laboratórios:
- Ômicron = 138 (41,57%)
- Delta = 194 (58,43%)
- Gama = 0
- Outras = 0
Com dados de: Fiocruz/RJ, UFSC, Dasa e Feevale
Para navegar, filtrar e interagir com algumas das informações apresentadas acima, clique aqui. O ITpS agradece aos pesquisadores e laboratórios que coletaram, geraram e submeteram os dados e metadados, aqui apresentados, para o banco de dados Gisaid.
We gratefully acknowledge all data contributors, i.e. the Authors and Originating labs, responsible for obtaining the specimens, and their Submitting labs for generating the genetic sequence and metadata and sharing via the GISAID Initiative (*), on which this research is based.
* Elbe, S., and Buckland -Merrett, G. (2017) Data, disease and diplomacy: GISAID's innovative contribution to global health. Global Challenges, 1:33-46. DOI: 10.1002/gch2.1018
https://onlinelibrary.wiley.com/doi/10.1002/gch2.1018
VOLTAR






